Co to jest GLM w R?
Uogólnione modele liniowe to podzbiór modeli regresji liniowej i skutecznie obsługują rozkłady niestandardowe. W tym celu zaleca się użycie funkcji glm (). GLM działa dobrze ze zmienną, gdy wariancja nie jest stała i rozkład normalny. Zdefiniowano funkcję łączenia w celu przekształcenia zmiennej odpowiedzi w celu dopasowania do odpowiedniego modelu. Model LM jest wykonywany zarówno z rodziny, jak i formuły. Model GLM ma trzy kluczowe komponenty zwane losowymi (prawdopodobieństwo), systematyczne (predyktor liniowy), komponent łącza (dla funkcji logit). Zaletą używania glm jest to, że mają elastyczność modelu, nie wymagają stałej wariancji, a ten model pasuje do oszacowania maksymalnego prawdopodobieństwa i jego współczynników. W tym temacie poznamy GLM w języku R.
Funkcja GLM
Składnia: glm (formuła, rodzina, dane, wagi, podzbiór, Start = null, model = PRAWDA, metoda = ””…)
Typy rodziny (w tym typy modeli) obejmują dwumianowy, Poissona, Gaussa, gamma, quasi. Każda dystrybucja ma inne zastosowanie i może być używana w klasyfikacji i prognozach. A gdy model jest gaussowski, odpowiedź powinna być prawdziwą liczbą całkowitą.
A gdy model jest dwumianowy, odpowiedzią powinny być klasy z wartościami binarnymi.
A gdy modelem jest Poissona, odpowiedź powinna być nieujemna z wartością liczbową.
A gdy modelem jest gamma, odpowiedź powinna być dodatnią wartością liczbową.
glm.fit () - Aby dopasować model
Lrfit () - oznacza dopasowanie regresji logistycznej.
update () - pomaga w aktualizacji modelu.
anova () - jest to test opcjonalny.
Jak utworzyć GLM w R?
Zobaczymy, jak stworzyć łatwy uogólniony model liniowy z danymi binarnymi za pomocą funkcji glm (). I kontynuując zestaw danych Drzewa.
Przykłady
// Importowanie bibliotekilibrary(dplyr)
glimpse(trees)

Aby zobaczyć wartości jakościowe, przypisane są czynniki.
levels(factor(trees$Girth))
// Weryfikacja zmiennych ciągłych
library(dplyr)
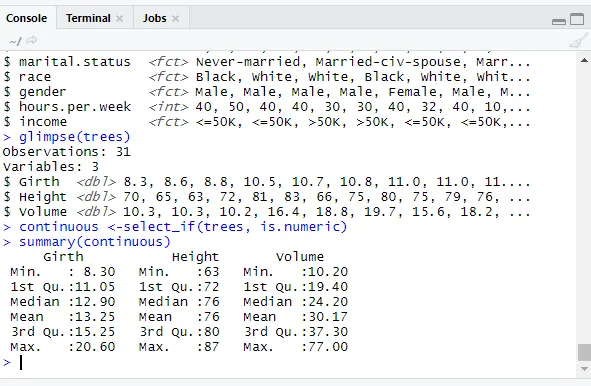
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Dołączanie drzewa danych do wyszukiwania R Pathattach (drzewa)
x<-glm(Volume~Height+Girth)
x
Wynik:
| Zadzwoń: glm (formuła = objętość ~ wysokość + obwód)
Współczynniki: (Przechwyć) Obwód wysokości -57, 9877 0, 3393 4, 7082 Stopnie swobody: 30 ogółem (tj. Zero); 28 Resztki Null Deviance: 8106 Odchylenie resztkowe: 421, 9 AIC: 176, 9 |
summary(x)
| Połączenie:
glm (formuła = objętość ~ wysokość + obwód) Pozostałości odchyleń: Min 1Q Mediana 3Q Max -6, 4065 -2, 6449 -0, 2876 2.2003 8, 4847 Współczynniki: Oszacuj Std. Błąd wartość t Pr (> | t |) (Przechwyć) -57, 9877 8, 6382 -6, 713 2, 75e-07 *** Wysokość 0, 3393 0, 1302 2, 607 0, 0145 * Obwód 4, 7082 0, 2643 17, 816 <2e-16 *** - Signif. kody: 0 „***” 0, 001 ”**„ 0, 01 ”*„ 0, 05 ”.” 0, 1 '' 1 (Parametr dyspersji dla rodziny gaussowskiej przyjęto jako 15.06862) Odchylenie zerowe: 8106, 08 przy 30 stopniach swobody Odchylenie resztkowe: 421, 92 na 28 stopniach swobody AIC: 176, 91 Liczba iteracji Fishera: 2 |
Dane wyjściowe funkcji podsumowania podaje wywołania, współczynniki i wartości resztkowe. Z powyższej odpowiedzi wynika, że zarówno współczynnik wzrostu, jak i obwód są nieistotne, ponieważ prawdopodobieństwo ich wystąpienia jest mniejsze niż 0, 5. I istnieją dwa warianty dewiacji o nazwach null i residual. Wreszcie, punktacja Fishera jest algorytmem, który rozwiązuje problemy z maksymalnym prawdopodobieństwem. W przypadku dwumianu odpowiedzią jest wektor lub macierz. cbind () służy do wiązania wektorów kolumnowych w macierzy. Aby uzyskać szczegółowe informacje na temat podsumowania dopasowania, wykorzystano.
Aby wykonać test typu kaptur, wykonywany jest następujący kod.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
Model pasuje
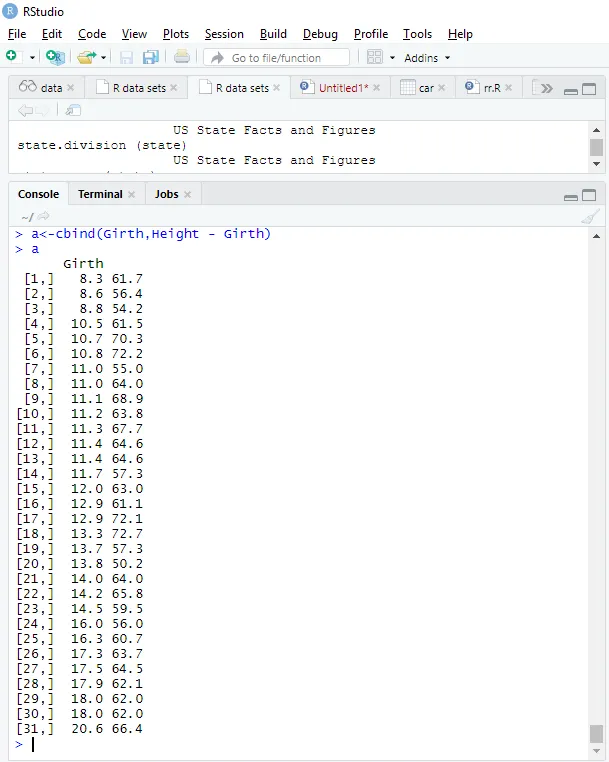
a<-cbind(Height, Girth - Height)
> a
podsumowanie (drzewa)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
Aby uzyskać odpowiednie odchylenie standardowe
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
Następnie odwołujemy się do zmiennej zliczania odpowiedzi w celu modelowania dobrego dopasowania odpowiedzi. Aby to obliczyć, wykorzystamy zestaw danych USAccDeath.
Wprowadźmy następujące fragmenty w konsoli R i zobaczmy, jak liczony jest rok i kwadrat roku.
data("USAccDeaths")
force(USAccDeaths)
// Analiza roku 1973–1978.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Połączenie:
glm (formuła = liczba ~ rok + rokSqr, rodzina = „poisson”, dane = dysk) Pozostałości odchyleń: Min 1Q Mediana 3Q Max -22, 4344 -6, 4401 -0, 0981 6, 0508 21, 4578 Współczynniki: Oszacuj Std. Błąd z wartością Pr (> | z |) (Przechwyć) 9, 187e + 00 3, 557e-03 2582.49 <2e-16 *** rok -7.207e-03 2.354e-04 -30.62 <2e-16 *** rok Sqr 8, 841e-05 3, 221e-06 27, 45 <2e-16 *** - Signif. kody: 0 „***” 0, 001 ”**„ 0, 01 ”*„ 0, 05 ”.” 0, 1 '' 1 (Parametr dyspersji dla rodziny Poissona przyjęty jako 1) Odchylenie zerowe: 7357, 4 na 71 stopniach swobody Odchylenie resztkowe: 6358, 0 na 69 stopniach swobody AIC: 7149, 8 Liczba iteracji Fishera: 4 |
Aby sprawdzić najlepsze dopasowanie modelu, można znaleźć następujące polecenie
resztki do testu. Z poniższego wyniku wartość wynosi 0.
1 - pchisq(deviance(a1), df.residual(a1))
Wykorzystanie rodziny QuasiPoisson dla większej wariancji podanych danych
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Połączenie:
glm (formuła = liczba ~ rok + rokSqr, rodzina = „quasipoisson”, dane = dysk) Pozostałości odchyleń: Min 1Q Mediana 3Q Max -22, 4344 -6, 4401 -0, 0981 6, 0508 21, 4578 Współczynniki: Oszacuj Std. Błąd wartość t Pr (> | t |) (Przechwyć) 9, 187e + 00 3, 417e-02 268, 822 <2e-16 *** rok -7.207e-03 2.261e-03 -3.188 0, 00216 ** rok Sqr 8, 841e-05 3, 095e-05 2, 857 0, 00565 ** - (Parametr dyspersji dla rodziny quasipoisson przyjęto jako 92, 28857) Odchylenie zerowe: 7357, 4 na 71 stopniach swobody Odchylenie resztkowe: 6358, 0 na 69 stopniach swobody AIC: NA Liczba iteracji Fishera: 4 |
Porównanie Poissona z dwumianową wartością AIC różni się znacznie. Można je analizować według dokładności i współczynnika przywołania. Następnym krokiem jest sprawdzenie, czy wariancja reszt jest proporcjonalna do średniej. Następnie możemy wykreślić za pomocą biblioteki ROCR, aby ulepszyć model.
Wniosek
Dlatego skupiliśmy się na specjalnym modelu zwanym uogólnionym modelem liniowym, który pomaga w skupieniu i oszacowaniu parametrów modelu. Jest to przede wszystkim potencjał zmiennej zmiennej odpowiedzi ciągłej. I widzieliśmy, jak glm pasuje do wbudowanych pakietów R. Są to najpopularniejsze podejścia do pomiaru danych zliczeniowych oraz niezawodne narzędzie do technik klasyfikacji stosowanych przez naukowca danych. Język R oczywiście pomaga w wykonywaniu skomplikowanych funkcji matematycznych
Polecane artykuły
To jest przewodnik po GLM w R. Tutaj omawiamy funkcję GLM i sposób tworzenia GLM w R z przykładowymi zestawami danych i danymi wyjściowymi. Możesz także spojrzeć na następujący artykuł, aby dowiedzieć się więcej -
- R Język programowania
- Architektura Big Data
- Regresja logistyczna w R.
- Zadania analityki Big Data
- Regresja Poissona w R | Wdrażanie regresji Poissona